Romana Ďuráčiová, studentka FIT VUT, získala dvě prestižní ocenění za vývoj a prezentaci grafického prostředí Foldify na studentské konferenci Excel@FIT 2025. Kristián Kováč prezentoval projekt Genomic Data Infrastructure (GDI) zaměřený na bezpečné zpracování a analýzu citlivých genomických dat v prostředí mezinárodní spolupráce. Oba projekty jsou řešeny v Centru CERIT-SC pod vedením Lukáše Hejtmánka.
 S potěšením oznamujeme, že Romana Ďuráčiová, studentka Fakulty informačních technologií VUT, získala dvě prestižní ocenění za vývoj a prezentaci grafického prostředí Foldify na studentské konferenci Excel@FIT 2025. Tento nástroj, který vzniká v centru CERIT-SC pod vedením Lukáše Hejtmánka, byl oceněn odbornou porotou i partnery z průmyslu.
S potěšením oznamujeme, že Romana Ďuráčiová, studentka Fakulty informačních technologií VUT, získala dvě prestižní ocenění za vývoj a prezentaci grafického prostředí Foldify na studentské konferenci Excel@FIT 2025. Tento nástroj, který vzniká v centru CERIT-SC pod vedením Lukáše Hejtmánka, byl oceněn odbornou porotou i partnery z průmyslu.
Foldify je intuitivní grafické prostředí určené pro práci s nástrojem AlphaFold, revoluční technologií pro předpovídání prostorové struktury proteinů. AlphaFold, vyvinutý společností DeepMind, přinesl v roce 2020 zásadní průlom díky verzi AlphaFold2, která dosáhla mimořádné přesnosti v předpovědích proteinových struktur, a to na úrovni srovnatelné s experimentálními metodami, jako je rentgenová krystalografie. Tato technologie byla označena za jeden z největších pokroků v biologii a přispěla k udělení Nobelovy ceny za chemii týmu, který se podílel na jejím vývoji.
Na AlphaFold2 navazuje AlphaFold3, představený v roce 2024, který dále rozšiřuje možnosti predikce na celé biomolekulární komplexy. Kromě proteinů dokáže modelovat i interakce s DNA, RNA, ligandy a ionty, což otevírá nové cesty pro vývoj léčiv, porozumění regulačním mechanismům v buňkách nebo návrh syntetických biomolekul. AlphaFold3 je výsledkem spolupráce DeepMind a Isomorphic Labs a představuje nový standard v oblasti strukturální biologie.
Motivací k vytvoření Foldify byla potřeba zpřístupnit revoluční technologii AlphaFold širší vědecké komunitě. Použití AlphaFold je pro běžné biology často komplikované, protože vyžaduje znalost práce s příkazovou řádkou a přístup k výpočetně náročné infrastruktuře. Foldify na tento problém reaguje vytvořením uživatelsky přívětivého grafického rozhraní, které umožňuje jednoduché zadávání úloh a vizualizaci výsledků bez nutnosti technických znalostí, a zároveň napojuje uživatele na výkonný výpočetní cluster provozovaný CERIT-SC.
Foldify běží v prostředí Kubernetes provozovaném CERIT-SC a je navrženo tak, aby zjednodušilo a zpřístupnilo práci s predikcí proteinových struktur. Nabízí jednoduché zadávání výpočtů, zpětnou vazbu, vizualizaci výsledků a možnost stažení predikovaných proteinů. Obsahuje rovněž optimalizované nástroje AlphaFold, ColabFold, OmegaFold, ESMFold. Za pět měsíců provozu bylo úspěšně dokončeno více než 700 predikcí.
Webové rozhraní: https://foldify.cloud.e-infra.cz/
Návod: https://docs.cerit.io/en/docs/web-apps/foldify
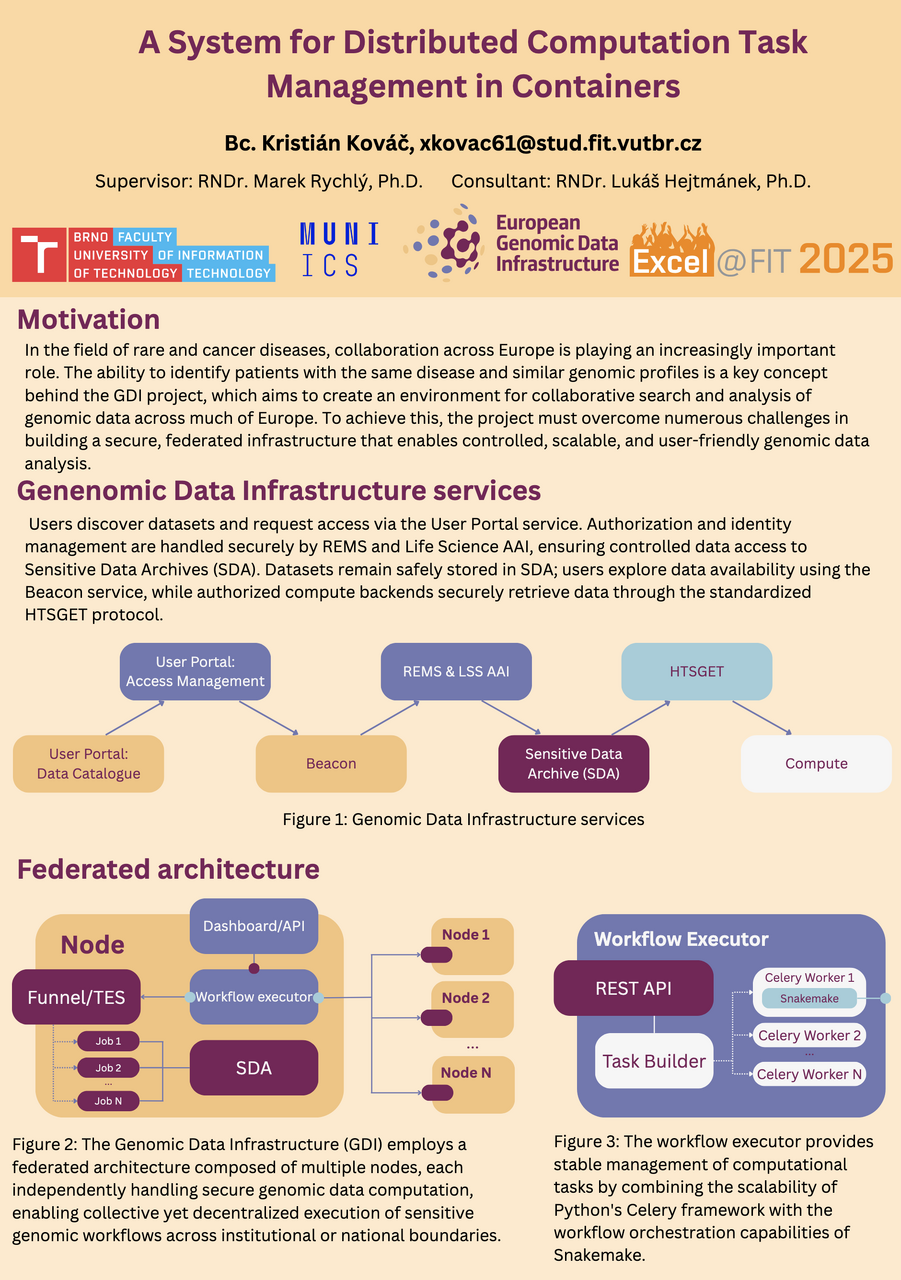 Kromě Romany se konference zúčastnil také další student FIT VUT, Kristián Kováč, který prezentoval projekt Genomic Data Infrastructure (GDI) zaměřený na bezpečné zpracování a analýzu citlivých genomických dat v prostředí mezinárodní spolupráce. Přestože nezískal cenu, jeho přínos pro náš tým je mimořádně významný.
Kromě Romany se konference zúčastnil také další student FIT VUT, Kristián Kováč, který prezentoval projekt Genomic Data Infrastructure (GDI) zaměřený na bezpečné zpracování a analýzu citlivých genomických dat v prostředí mezinárodní spolupráce. Přestože nezískal cenu, jeho přínos pro náš tým je mimořádně významný.
Kristian, pod vedením Lukáše Hejtmánka z centra CERIT-SC, vyvíjí komplexní portál a infrastrukturu pro podporu bezpečného workflow, včetně komponent pro vkládání dat, správu přístupových oprávnění a samotnou analýzu.
Infrastruktura implementuje moderní přístupy jako OAuth2, GA4GH Visas, a využívá nástroje jako Snakemake, Kubernetes a Celery pro škálovatelné a robustní workflow. Výsledkem je federovaná platforma, která umožňuje analýzu genomických dat napříč institucemi při zachování vysoké úrovně bezpečnosti a kontroly přístupu.
Architektura systému je decentralizovaná – jednotlivé výpočetní uzly operují nezávisle a uchovávají citlivá data lokálně ve svých bezpečných úložištích SDA. Každý uzel obsahuje tzv. Workflow Executor, který koordinuje běh výpočetních úloh. Zpracování dat se odehrává výhradně uvnitř autorizovaných služeb, což umožňuje spouštět workflow napříč institucemi bez kompromitace dat. Při každém spuštění úlohy je zajištěna autentizace tokenem, což umožňuje transparentní práci s citlivými daty. Celkově systém poskytuje robustní, škálovatelné a bezpečné prostředí pro spolupráci v oblasti genomických výpočtů.
Projekt GDI tak významně přispívá k realizaci cílů mezinárodních iniciativ jako B1MG (Beyond 1 Million Genomes) a 1+MG, které usilují o propojení genomických dat napříč Evropou pro účely výzkumu a personalizované medicíny.
O projektu: https://gdi.onemilliongenomes.eu/

